マルチプルアライメントと分子系統樹実習
-
課題シート
- 準備
まず、以下の三つのプログラムをダウンロードし、デスクトップにコピーする
- マルチプルアライメントのプログラム:ClustalX ver 1.83 (Windows版)
- 有根系統樹の描画プログラム:njplot(Windows版)
- 無根系統樹の描画プログラム:unrooted(Windows版)
- マルチプルアライメントのプログラム:ClustalX ver 1.83 (Windows版)
- 分子系統樹から生物種の系統関係を推定する
本演習ではミトコンドリアのCytochrome bのアミノ酸配列の分子系統樹を描くことにより、生物種の 系統関係を推定する。 まず、以下の三つのファイルをダウンロードする。- 魚類の配列+外群としてハエ(DROME)の配列:cyb_fish.seq :演習3
- 哺乳類の配列+外群としてニワトリ(CHICK)の配列:cyb_mammal.seq :演習4
- 脊椎動物の配列+外群としてハエ(DROME)の配列:cyb_verte.seq :演習5
あとは、それぞれの配列について以下の手順で、系統樹を作成する。
- clustalxのアイコンをクリックして起動する
- [File]メニューから[Load Sequence]を選び、配列のファイル(たとえばcyb_fish.seq)を読み込む
- [Alignment]メニューから[Do Complete Alignment]を選び、マルチプルアライメントを実行する
- [Tree]メニューから[Draw N-J Tree]あるいは[Bootstrap N-J Tree]を選び、系統樹を作成する
- njplotのアイコンをクリックして起動する
- [File]メニューから[Open]を選び、ClustalXで出力された系統樹のファイル(*.phか*.phb)を 読み込む
- [New Outgroup]ボタンを押し、適当な外群を選択する
- 魚類の配列+外群としてハエ(DROME)の配列:cyb_fish.seq :演習3
- 生物種を選択し、ヘモグロビンα鎖の配列を用いて系統関係を推定する
演習6
本演習では系統関係を調べたい10種前後の生物種をヘモグロビンα鎖の配列がわかっている生物種のリストから選択し、外群を加えた、アミノ酸配列のファイルを作成する。作成したファイルについてこれまでと同様、マルチプルアラインメントを行い、系統樹を作成する まずリストから任意の生物種を選択し、その5文字の生物種コードをリストアップする。それぞれの生物種名コードのまえにヘモグロビンα鎖を表すHBA_を加える。例えば、ヒトの場合には生物種コードはHUMANであるから、タンパク質名としてHBA_HUMANをスイスプロットのホームページで検索する、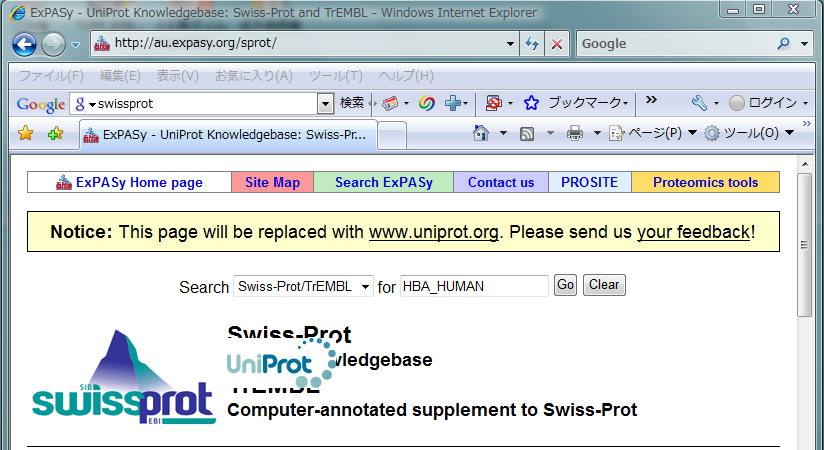
クェリー欄に "HBA_HUMAN" とタイプし "Go" ボタンを押す。
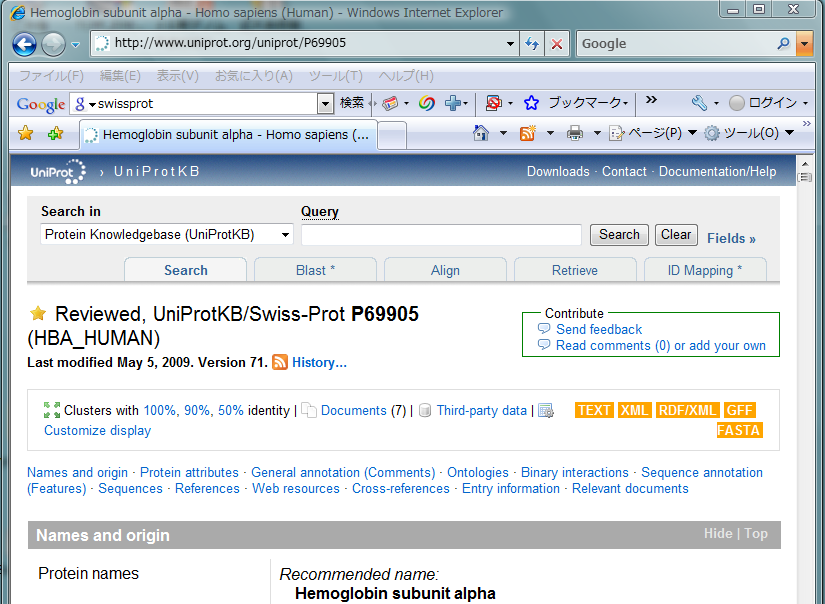
右側の黄色い "FASTA" というボタンを押し ワードパッド画面上の文字列を選択し、コピーして、 メモ帳アプリケーション上に貼付けてゆく。
このとき、各エントリの ">" ではじまるタイトル行のはじめに、英語名またはローマ字でわかり易い生物種名を記入しておくとよい、
例)
>sp|P69905|HBA_HUMAN Hemoglobin subunit alpha OS=Homo sapiens GN=HBA1 PE=1 SV=2
MVLSPADKTNVKAAWGKVGAHAGEYGAEALERMFLSFPTTKTYFPHFDLSHGSAQVKGHG
KKVADALTNAVAHVDDMPNALSALSDLHAHKLRVDPVNFKLLSHCLLVTLAAHLPAEFTP
AVHASLDKFLASVSTVLTSKYRを
>Hito sp|P69905|HBA_HUMAN Hemoglobin subunit alpha OS=Homo sapiens GN=HBA1 PE=1 SV=2
MVLSPADKTNVKAAWGKVGAHAGEYGAEALERMFLSFPTTKTYFPHFDLSHGSAQVKGHG
KKVADALTNAVAHVDDMPNALSALSDLHAHKLRVDPVNFKLLSHCLLVTLAAHLPAEFTP
AVHASLDKFLASVSTVLTSKYRとしておくと後で結果が見やすくなる。
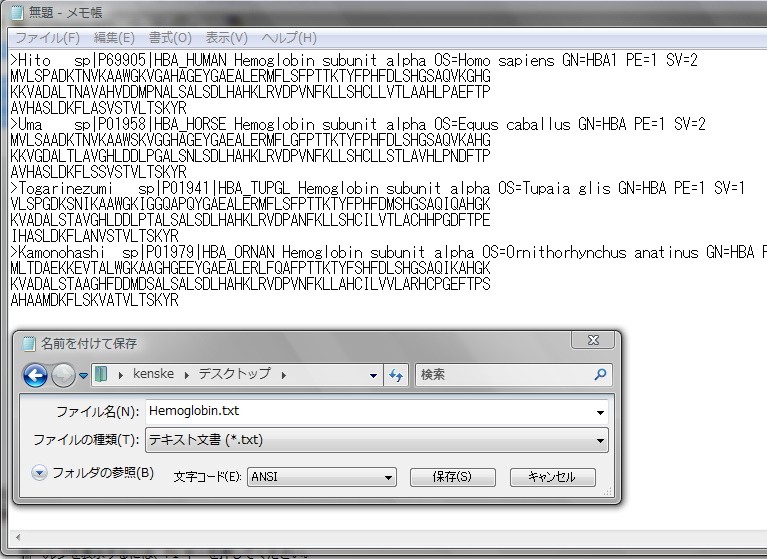
このようにして10前後の選択した生物種のヘモグロビンα鎖(HBA_)配列を並べたものを作成し、適切な名前(たとえば"Hemoglobin.seq")を付けて保存する。
作成したアミノ酸配列ファイルについて clustalx を用いてマルチプルアラインメントをおこない、 unrootedの系統樹を描画して演習用紙にコメントを記入する。